

诊断学理论与实践 ›› 2022, Vol. 21 ›› Issue (04): 456-461.doi: 10.16150/j.1671-2870.2022.04.007
收稿日期:2022-08-23
出版日期:2022-08-25
发布日期:2022-11-07
通讯作者:
何瑾
E-mail:hejinfjmu@hotmail.com
YANG Hui1, LI Yunlu2, YANG Kang2, LI Shiju1, HE Jin2( )
)
Received:2022-08-23
Online:2022-08-25
Published:2022-11-07
Contact:
HE Jin
E-mail:hejinfjmu@hotmail.com
摘要:
目的:分析近亲家系全外显子测序(whole exome sequencing, WES)中漏诊5例进行性肌阵挛共济失调(progressive myoclonic ataxia, PMA)患者的原因,并探讨对策。方法:收集5例WES未发现致病突变而临床拟诊为PMA患者,对其近亲婚配家系(三代以内旁系血亲婚配)进行全基因组SNP芯片扫描,通过纯合子定位技术及单倍型分析定位致病区间,结合患者的肌阵挛和共济失调表型,明确其可能的致病基因。采用Sanger测序对候选的突变位点进行家系共分离分析,最后通过溯源WES比对过程中的SAM与BAM文件,分析WES漏诊的原因。结果:纯合区间定位和重分析WES发现,5例PMA家系为NEU1基因c. 544A>G(p.S182G)纯合突变导致的Ⅰ型唾液酸沉积症(sialidosis type Ⅰ, ST-Ⅰ)。人类基因组参考序列中的ALT contig在WES比对过程中可造成包括NEU1基因在内的多个PMA致病基因检出遗漏。结论:临床诊断中,常染色体隐性遗传的PMA需关注NEU1基因突变所导致的ST-Ⅰ。对于WES未发现致病位点的PMA家系,可结合家系遗传方式、全基因组SNP芯片等方法定位致病区间,再结合患者的临床表型以明确其疾病致病基因。
中图分类号:
杨慧, 李云璐, 杨康, 李世举, 何瑾. 进行性肌阵挛共济失调近亲家系全外显子测序漏诊原因分析与对策[J]. 诊断学理论与实践, 2022, 21(04): 456-461.
YANG Hui, LI Yunlu, YANG Kang, LI Shiju, HE Jin. Misdiagnosis by whole exome sequencing in progressive myoclonic ataxia consanguineous families: causes and strategies[J]. Journal of Diagnostics Concepts & Practice, 2022, 21(04): 456-461.
表1
5个近亲结婚PMA家系的临床表型归纳
| 家系 | 患者 | 性别 | 就诊年龄(岁) | 发病年龄(岁) | 首发症状 | 肌阵挛 | 共济失调 | 癫痫 | 认知 | |
|---|---|---|---|---|---|---|---|---|---|---|
| 发病年龄(岁) | 持续发作 | |||||||||
| 1 | Ⅱ-2 | 女 | 36 | 12 | 共济失调 | 17 | - | + | + | - |
| Ⅱ-3 | 女 | 35 | 14 | 共济失调 | 15 | - | + | - | - | |
| 2 | Ⅱ-2 | 女 | 40 | 17 | 肌阵挛 | 17 | + | + | + | - |
| Ⅱ-4 | 男 | 36 | 15 | 共济失调 | 17 | - | + | + | - | |
| 3 | Ⅱ-1 | 男 | 38 | 21 | 肌阵挛 | 21 | - | + | + | - |
| Ⅱ-2 | 男 | 34 | 17 | 癫痫 | 17 | + | + | + | - | |
| 4 | Ⅱ-1 | 女 | 40 | 18 | 肌阵挛 | 18 | - | + | - | - |
| Ⅱ-2 | 女 | 38 | 16 | 肌阵挛 | 16 | - | + | + | - | |
| 5 | Ⅱ-2 | 男 | 43 | 13 | 肌阵挛 | 13 | + | + | + | - |
| Ⅱ-3 | 男 | 30 | 19 | 口吃 | 20 | - | + | - | - | |

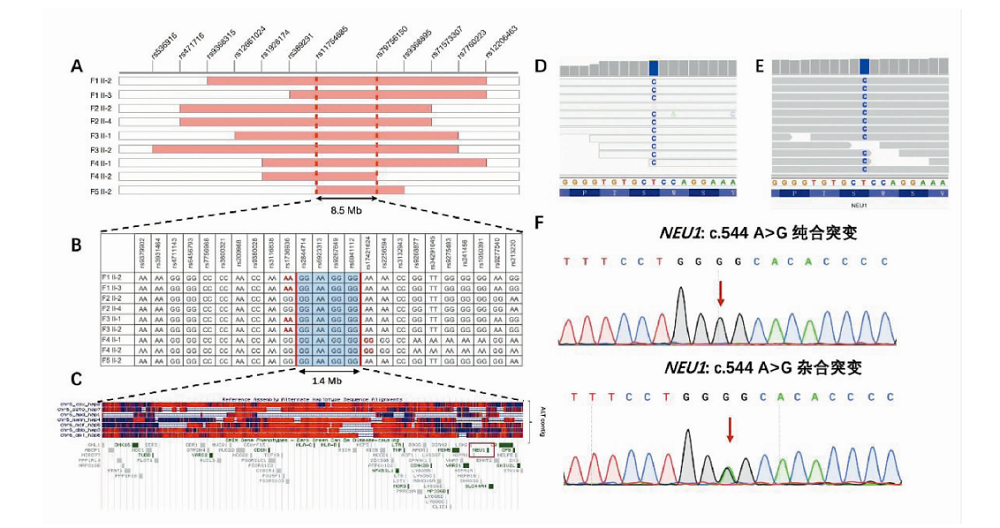
图2
5个近亲结婚PMA家系纯合区间定位、单倍型分析、全外数据重分析以及致病位点检测 A:5例PMA家系纯合区间定位,明确5个家系共享rs11754685和rs79756150范围内8.5Mb纯合区间;B:5例PMA家系在rs2844714和rs6941114范围内1.4 Mb区间内共享同一个单倍型;C:1.4 Mb区间内hg19人类参考基因组,NEU1基因部分序列与ALT contig呈现高度同源性;D:ALT contig序列参与比对造成的IGV视图中异常的灰框白底序列;E:去除ALT contig序列参与比对后IGV视图中正常的灰底序列;F:NEU1基因Sanger测序图,c.544A>G纯合突变为患者,c.544A>G杂合突变为携带者。
| [1] |
Muona M, Berkovic SF, Dibbens LM, et al. A recurrent de novo mutation in KCNC1 causes progressive myo-clonus epilepsy[J]. Nat Genet, 2015, 47(1):39-46.
doi: 10.1038/ng.3144 |
| [2] |
Marseille Consensus Group. Classification of progressive myoclonus epilepsies and related disorders[J]. Ann Neurol, 1990, 28(1):113-116.
doi: 10.1002/ana.410280129 URL |
| [3] |
van der Veen S, Zutt R, Elting JWJ, et al. Progressive myoclonus ataxia: time for a new definition?[J]. Mov Disord, 2018, 33(8):1281-1286.
doi: 10.1002/mds.27412 URL |
| [4] |
Rossi M, van der Veen S, Merello M, et al. Myoclonu-sataxia syndromes: a diagnostic approach[J]. Mov Disord Clin Pract, 2020, 8(1):9-24.
doi: 10.1002/mdc3.13106 URL |
| [5] |
Kherraf ZE, Cazin C, Bouker A, et al. Whole-exome sequencing improves the diagnosis and care of men with non-obstructive azoospermia[J]. Am J Hum Genet, 2022, 109(3):508-517.
doi: 10.1016/j.ajhg.2022.01.011 URL |
| [6] |
Gabriel H, Korinth D, Ritthaler M, et al. Trio exome sequencing is highly relevant in prenatal diagnostics[J]. Prenat Diagn, 2022, 42(7):845-851.
doi: 10.1002/pd.6081 URL |
| [7] | Alkuraya FS. Discovery of rare homozygous mutations from studies of consanguineous pedigrees[J]. Curr Protoc Hum Genet, 2012,Chapter 6:Unit6.12. |
| [8] |
Purcell S, Neale B, Todd-Brown K, et al. PLINK: a tool set for whole-genome association and population-based linkage analyses[J]. Am J Hum Genet, 2007, 81(3):559-575.
doi: 10.1086/519795 pmid: 17701901 |
| [9] |
Hamilton A, Tétreault M, Dyment DA, et al. Concordance between whole-exome sequencing and clinical Sanger sequencing: implications for patient care[J]. Mol Genet Genomic Med, 2016, 4(5):504-512.
doi: 10.1002/mgg3.223 URL |
| [10] |
Goodwin S, McPherson JD, McCombie WR. Coming of age: ten years of next-generation sequencing technologies[J]. Nat Rev Genet, 2016, 17(6):333-351.
doi: 10.1038/nrg.2016.49 pmid: 27184599 |
| [11] |
Ma X, Shao Y, Tian L, et al. Analysis of error profiles in deep next-generation sequencing data[J]. Genome Biol, 2019, 20(1):50.
doi: 10.1186/s13059-019-1659-6 pmid: 30867008 |
| [12] |
Ross MG, Russ C, Costello M, et al. Characterizing and measuring bias in sequence data[J]. Genome Biol, 2013, 14(5):R51.
doi: 10.1186/gb-2013-14-5-r51 URL |
| [13] |
Lelieveld SH, Spielmann M, Mundlos S, et al. Comparison of exome and genome sequencing technologies for the complete capture of protein-coding regions[J]. Hum Mutat, 2015, 36(8):815-822.
doi: 10.1002/humu.22813 pmid: 25973577 |
| [14] |
Kong SW, Lee IH, Liu X, et al. Measuring coverage and accuracy of whole-exome sequencing in clinical context[J]. Genet Med, 2018, 20(12):1617-1626.
doi: 10.1038/gim.2018.51 pmid: 29789557 |
| [15] |
Reis CS, Quental S, Fernandes S, et al. Whole-exome sequencing targeting a gene panel for sensorineural hearing loss: the first portuguese cohort study[J]. Cytogenet Genome Res, 2022, 162(1-2):1-9.
doi: 10.1159/000523840 URL |
| [16] | Haarman AEG, Thiadens AAHJ, van Tienhoven M, et al. Whole exome sequencing of known eye genes reveals genetic causes for high myopia[J/OL]. Hum Mol Genet, 2022[2022-08-23]. https://pubmed.ncbi.nlm.nih.gov/35567543/. |
| [17] |
Sreenivasan R, Bell K, van den Bergen J, et al. Whole exome sequencing reveals copy number variants in individuals with disorders of sex development[J]. Mol Cell Endocrinol, 2022, 546:111570.
doi: 10.1016/j.mce.2022.111570 URL |
| [18] |
Mandelker D, Schmidt RJ, Ankala A, et al. Navigating highly homologous genes in a molecular diagnostic setting: a resource for clinical next-generation sequencing[J]. Genet Med, 2016, 18(12):1282-1289.
doi: 10.1038/gim.2016.58 pmid: 27228465 |
| [19] |
Cao YN, Li L, Xu M, et al. The ChinaMAP analytics of deep whole genome sequences in 10 588 individuals[J]. Cell Res, 2020, 30(9):717-731.
doi: 10.1038/s41422-020-0322-9 URL |
| [1] | 何新, 陈慧, 冯炜炜. 机器学习算法在辅助超声诊断附件肿块良恶性中的应用研究进展[J]. 诊断学理论与实践, 2022, 21(04): 541-546. |
| [2] | 王文涵, 夏蜀珺, 詹维伟. 长链非编码RNA ENST00000489676在超声评估甲状腺乳头状癌颈部淋巴结转移中的应用[J]. 诊断学理论与实践, 2022, 21(04): 514-519. |
| [3] | 顾炫, 柳俊. 超声筛查鉴别胰腺实性假乳头状瘤与胰腺导管腺癌的研究分析[J]. 诊断学理论与实践, 2022, 21(04): 504-508. |
| [4] | 马雪菲, 王学锋, 王侃侃. 浆细胞瘤变异体易位1和MYC基因在泛癌中的表达及生存期预测价值分析[J]. 诊断学理论与实践, 2022, 21(04): 490-496. |
| [5] | 屈骞, 海汪溪, 胡生焰, 张敏, 陈肖玥, 周熠磊, 王瑾, 胡晓平, 李彪, 胡佳佳. 多巴胺转运蛋白显像探针18F-FP-CIT的AllinOne模块自动化制备及大鼠基底节Micro PET/CT显像[J]. 诊断学理论与实践, 2022, 21(04): 482-489. |
| [6] | 车稳, 柳蒋书, 陈晓炎, 王朝夫, 袁菲, 王璇. 肺混合性鳞状细胞和腺性乳头状瘤2例临床病理特征及冷冻切片病理诊断误诊分析[J]. 诊断学理论与实践, 2022, 21(04): 476-481. |
| [7] | 徐程, 徐欣欣, 田烨, 范嘉盈, 宋珍, 杨玲. 下呼吸道流感嗜血杆菌定植通过Toll样受体4影响哮喘小鼠免疫失衡[J]. 诊断学理论与实践, 2022, 21(04): 470-475. |
| [8] | 鲍萍萍, 吴春晓, 顾凯, 庞怡, 王春芳, 施亮, 向詠梅, 龚杨明, 窦剑明, 吴梦吟, 付晨, 施燕. 上海市2016年胃癌发病特征及2002年至2016年胃癌发病趋势分析[J]. 诊断学理论与实践, 2022, 21(04): 462-469. |
| [9] | 胡静静, 吕海伟, 荀静娜, 沈银忠, 刘莉, 卢洪洲. 2012年至2021年上海市艾滋病合并分枝杆菌感染患者的菌种分布特征[J]. 诊断学理论与实践, 2022, 21(04): 450-455. |
| [10] | 陈志敏, 刘波, 何浩岚, 何耀祖, 冯理智, 刘新华, 张坚生, 蔡卫平, 李凌华. 133例艾滋病合并马尔尼菲篮状菌病死亡病例分析[J]. 诊断学理论与实践, 2022, 21(04): 444-449. |
| [11] | 杜燕然, 焦景, 任芸芸, 周建桥. 超声影像组学技术在评估胎肺成熟度中的应用[J]. 诊断学理论与实践, 2022, 21(03): 326-330. |
| [12] | 张娟娟, 何亲羽, 杨媛艳, 董治亚, 肖园, 陈立芬, 张彩萍. Lamb-Shaffer综合征表现为矮小症伴语言、运动发育迟缓一例及文献复习[J]. 诊断学理论与实践, 2022, 21(03): 336-342. |
| [13] | 刘欣, 綦才辉, 王振竞, 吕娜, 王少婷, 王淑萍. 胰高血糖素样肽-1激动剂Exendin-4 刺激小鼠胚胎成骨细胞前体细胞MC3T3-E1的转录组学体外研究[J]. 诊断学理论与实践, 2022, 21(03): 367-373. |
| [14] | 阮玉凤, 胡丽萍, 陈史蓉, 尹君, 孙璟. 上海浦东新区全科医师对规范化诊治幽门螺杆菌感染掌握程度的调研[J]. 诊断学理论与实践, 2022, 21(03): 399-404. |
| [15] | 马少辰, 郭昕, 王铭维, 王惠君, 余启军, 苏文月, 王华龙, 马芹颖. 基于游戏的脑电神经反馈训练对认知功能改善作用的研究[J]. 诊断学理论与实践, 2022, 21(01): 41-45. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||