

诊断学理论与实践 ›› 2022, Vol. 21 ›› Issue (05): 567-574.doi: 10.16150/j.1671-2870.2022.05.004
收稿日期:2022-09-13
出版日期:2022-10-25
发布日期:2023-01-29
通讯作者:
于颖彦
E-mail:yingyan3y@sjtu.edu.cn
基金资助:
YANG Ruixin, DU Yutong, YAN Ranlin, ZHU Zhenggang, LI Chen, YU Yingyan( )
)
Received:2022-09-13
Online:2022-10-25
Published:2023-01-29
Contact:
YU Yingyan
E-mail:yingyan3y@sjtu.edu.cn
摘要:
目的:探索在消化道上皮源性肿瘤单细胞转录组测序(简称单细胞测序)中提高组织单细胞悬液中上皮细胞占比的方法,以改善检测样本质量。方法:分析GEO公共数据库内常见消化道上皮源性肿瘤(胃癌、结肠直肠癌及胰腺癌)单细胞转录组测序数据集中的上皮细胞组分占比。比较传统酶消化法以及酶消化后辅以上机前机械性物理吹打法(改良法)处理后单细胞悬液中上皮细胞占比和细胞活力差异。结果:分析公共数据库内数据显示,4份正常胃上皮及胃癌样本的上皮细胞占比为17.05%、6.11%、8.93%和14.66%; 2份结肠直肠癌上皮细胞占比为3.86%和16.60%;2份胰腺癌上皮细胞占比为3.22%和21.37%。对2份新鲜采集的胃癌样本,采用传统酶消化法制备单细胞悬液的细胞结团率为56.26%±1.98%和38.34%±1.26%,细胞活力检测提示活细胞占比分别为98.43%±0.56%和97.24%±0.48%;而对传统酶消化法处理后的单细胞悬液在上机前用0.33 mm规格的胰岛素注射器施以机械性吹打操作30 s,可使单细胞悬液的上皮结团率降至22.78%±1.38%和14.46%±0.92%(P均<0.000 1);活细胞占比分别为95.16%±0.42%和93.52%±0.82%(P<0.05)。结论:在采用传统酶消化后辅以机械性物理吹打法的组织样本前处理操作,可减少上皮细胞结团,从而提高单细胞悬液中的上皮细胞占比,活细胞占比虽有小幅下降,但可满足上机要求。
中图分类号:
杨蕊馨, 杜宇童, 燕然林, 朱正纲, 李琛, 于颖彦. 消化道肿瘤单细胞转录组测序研究中生物样本前处理改良的探索[J]. 诊断学理论与实践, 2022, 21(05): 567-574.
YANG Ruixin, DU Yutong, YAN Ranlin, ZHU Zhenggang, LI Chen, YU Yingyan. Improving exploration of biological sample pretreatment in single-cell transcriptome sequencing of gastrointestinal tumors[J]. Journal of Diagnostics Concepts & Practice, 2022, 21(05): 567-574.
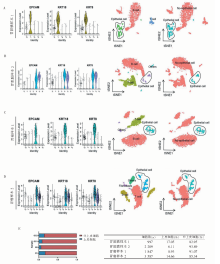
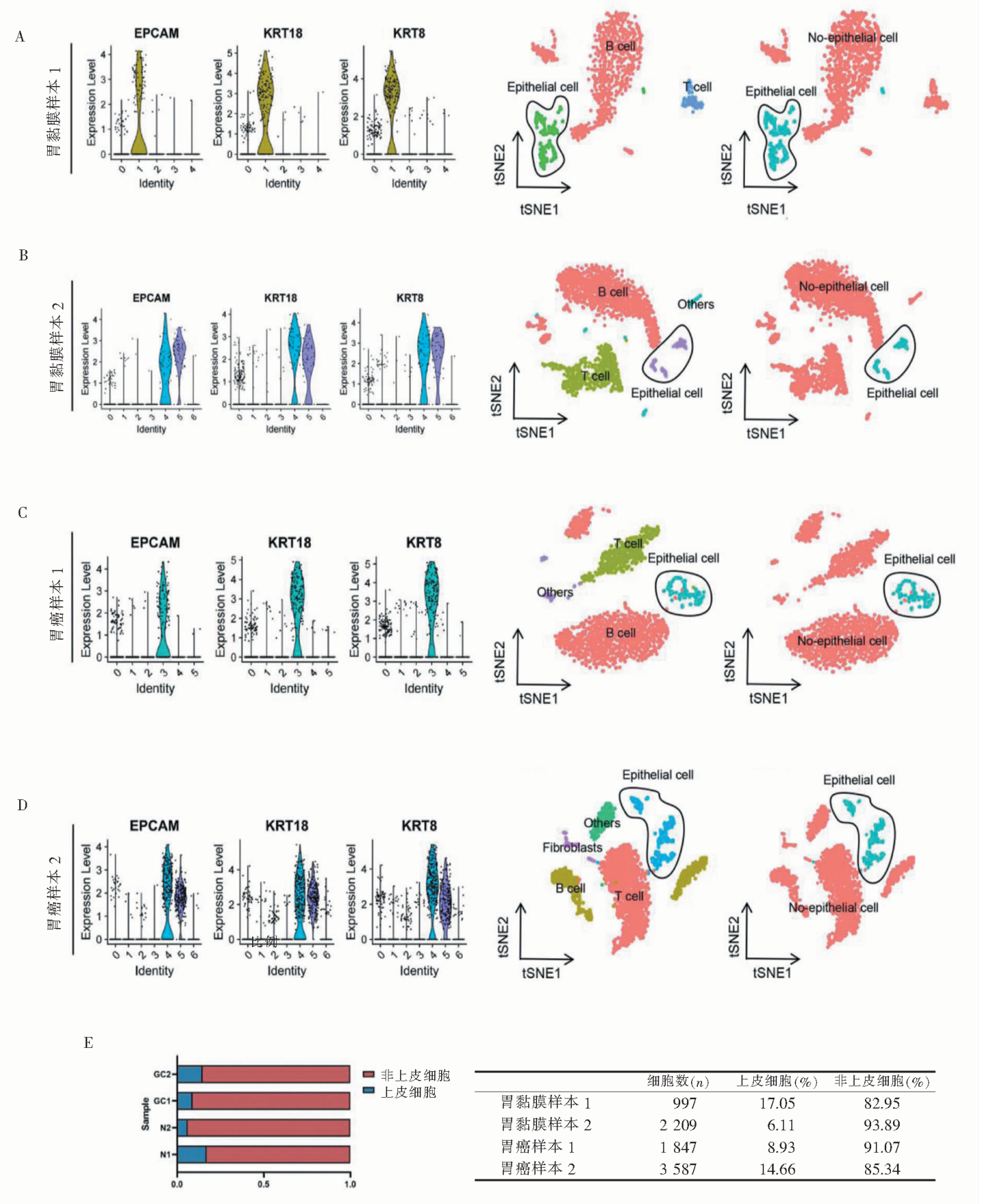
图1
胃上皮及胃癌数据集中细胞成分占比分析 注:B淋巴细胞群、T淋巴细胞群等统称为非上皮细胞群。A:胃黏膜样本1中的cluster 1群属于上皮细胞群,EPCAM、KRT18和KRT8呈高表达;右侧图显示上皮细胞占比(黑线圈出细胞群)并不高;B:胃黏膜样本2中的cluster 4、5为上皮细胞群,右侧图示上皮细胞占比较低(黑线圈出细胞群);C:胃癌样本1中的cluster 3为上皮细胞群,右侧图显示上皮细胞占比较低(黑线圈出细胞群);D:胃癌样本2中的cluster 4、5为上皮细胞群,右侧图示上皮细胞占比较低(黑线圈出细胞群);E. 胃黏膜样本1、胃黏膜样本2、胃癌样本1及胃癌样本2的单细胞测序数据集中上皮细胞成分和非上皮细胞成分占比详细情况。
| [1] |
Cancer Genome Atlas Research Network. Comprehensive molecular cha-racterization of gastric adenocarcinoma[J]. Nature, 2014, 513(7517):202-209.
doi: 10.1038/nature13480 URL |
| [2] |
Tang F, Barbacioru C, Wang Y, et al. mRNA-Seq whole-transcriptome analysis of a single cell[J]. Nat Methods, 2009, 6(5):377-382.
doi: 10.1038/nmeth.1315 pmid: 19349980 |
| [3] | Fu K, Hui B, Wang Q, et al. Single-cell RNA sequencing of immune cells in gastric cancer patients[J]. Aging (Albany NY), 2020, 12(3):2747-2763. |
| [4] |
Zhang Y, Song J, Zhao Z, et al. Single-cell transcriptome analysis reveals tumor immune microenvironment heterogenicity and granulocytes enrichment in colorectal cancer liver metastases[J]. Cancer Lett, 2020, 470:84-94.
doi: S0304-3835(19)30513-0 pmid: 31610266 |
| [5] |
Elyada E, Bolisetty M, Laise P, et al. Cross-species single-cell analysis of pancreatic ductal adenocarcinoma reveals antigen-presenting cancer-associated fibroblasts[J]. Cancer Discov, 2019, 9(8):1102-1123.
doi: 10.1158/2159-8290.CD-19-0094 pmid: 31197017 |
| [6] |
Zhang M, Hu S, Min M, et al. Dissecting transcriptional heterogeneity in primary gastric adenocarcinoma by single cell RNA sequencing[J]. Gut, 2021, 70(3):464-475.
doi: 10.1136/gutjnl-2019-320368 pmid: 32532891 |
| [7] |
Zhang P, Yang M, Zhang Y, et al. Dissecting the single-cell transcriptome network underlying gastric premalignant lesions and early gastric cancer[J]. Cell Rep, 2019, 27(6):1934-1947.
doi: S2211-1247(19)30525-X pmid: 31067475 |
| [8] |
Park J, Shrestha R, Qiu C, et al. Single-cell transcriptomics of the mouse kidney reveals potential cellular targets of kidney disease[J]. Science, 2018, 360(6390):758-763.
doi: 10.1126/science.aar2131 pmid: 29622724 |
| [9] |
Kim J, Park C, Kim KH, et al. Single-cell analysis of gastric pre-cancerous and cancer lesions reveals cell lineage diversity and intratumoral heterogeneity[J]. NPJ Precis Oncol, 2022, 6(1):9.
doi: 10.1038/s41698-022-00251-1 pmid: 35087207 |
| [10] |
Guo W, Zhang C, Wang X, et al. Resolving the difference between left-sided and right-sided colorectal cancer by single-cell sequencing[J]. JCI Insight, 2022, 7(1):e152616.
doi: 10.1172/jci.insight.152616 URL |
| [11] |
Lee JJ, Bernard V, Semaan A, et al. Elucidation of tumor-stromal heterogeneity and the ligand-receptor inte-ractome by single-cell transcriptomics in real-world pancreatic cancer biopsies[J]. Clin Cancer Res, 2021, 27(21):5912-5921.
doi: 10.1158/1078-0432.CCR-20-3925 URL |
| [12] |
Mei Y, Xiao W, Hu H, et al. Single-cell analyses reveal suppressive tumor microenvironment of human colorectal cancer[J]. Clin Transl Med, 2021, 11(6):e422.
doi: 10.1002/ctm2.422 pmid: 34185431 |
| [13] |
Zheng C, Zheng L, Yoo JK, et al. Landscape of infiltra-ting T cells in liver cancer revealed by single-cell sequencing[J]. Cell, 2017, 169(7):1342-1356,e16.
doi: 10.1016/j.cell.2017.05.035 URL |
| [14] |
Sathe A, Grimes SM, Lau BT, et al. Single-cell genomic characterization reveals the cellular reprogramming of the gastric tumor microenvironment[J]. Clin Cancer Res, 2020, 26(11):2640-2653.
doi: 10.1158/1078-0432.CCR-19-3231 pmid: 32060101 |
| [1] | 陈平, 徐莹, 吴云林. 消化内镜在早期胃癌诊断中的应用进展[J]. 诊断学理论与实践, 2022, 21(05): 551-554. |
| [2] | 王亚雷. 重视胃癌高危人群的内镜精查[J]. 诊断学理论与实践, 2022, 21(05): 555-559. |
| [3] | 马乾宸, 张本炎, 芮炜玮, 王婷, 罗方秀, 王朝夫, 袁菲. 中国3 071例胃癌病理分型分析[J]. 诊断学理论与实践, 2022, 21(05): 560-566. |
| [4] | 鲍萍萍, 吴春晓, 顾凯, 庞怡, 王春芳, 施亮, 向詠梅, 龚杨明, 窦剑明, 吴梦吟, 付晨, 施燕. 上海市2016年胃癌发病特征及2002年至2016年胃癌发病趋势分析[J]. 诊断学理论与实践, 2022, 21(04): 462-469. |
| [5] | 李娜娜, 齐涛, 朱黎明. 血清胃蛋白酶原、胃泌素17和幽门螺杆菌IgG抗体在胃部疾病初筛中的临床价值[J]. 诊断学理论与实践, 2022, 21(04): 509-513. |
| [6] | 杨翠萍, 杨晓金, 杨燕萍, 张梦茵, 谢玲, 俞骁珺, 蔡波尔, 陈登宇, 陈平, 吴云林. 人胃癌细胞BGC823中miR-200c靶基因产物波形蛋白的检测及功能研究[J]. 诊断学理论与实践, 2020, 19(04): 414-419. |
| [7] | 姜江, 曾志坤, 石博文, 潘召城, 颜凌, 王宇杰, 张欢. 玻璃动力学分析方法研究胃癌细胞运动学特点[J]. 诊断学理论与实践, 2019, 18(06): 645-648. |
| [8] | 王兰, 张欢, 葛颖倩, 陆静, 崔征, 颜凌, 潘自来. 胃癌肝转移病灶的人工智能辅助半自动分割软件的临床应用评估[J]. 诊断学理论与实践, 2019, 18(05): 515-520. |
| [9] | 周磊, 王虹, 徐慧明, 叶涛, 高建萍, 孙一骏, 谢军. 血清胃蛋白酶原对上海中心城区胃癌高危人群筛查慢性萎缩性胃炎的潜在价值[J]. 诊断学理论与实践, 2019, 18(05): 570-574. |
| [10] | 杨明鑫, 魏鸿擎, 谢静远. 单细胞转录组测序及其在肾脏疾病研究中的应用进展[J]. 诊断学理论与实践, 2019, 18(04): 475-478. |
| [11] | 张华, 李永兴, 乐嫣, 王文毓, 项明洁. 血清骨桥蛋白和组织多肽特异抗原联合检测在胃癌辅助诊断中的临床应用[J]. 诊断学理论与实践, 2018, 17(04): 428-432. |
| [12] | 武新洋, 张欢, 潘自来, 谭晶文, 杲霄源. 双源CT对原发性胃淋巴瘤和进展期胃癌的鉴别诊断价值[J]. 诊断学理论与实践, 2018, 17(01): 60-65. |
| [13] | 乔长婷, 李蕾, 邬安妮, 袁菲. 进展期胃癌人表皮生长因子受体2蛋白表达与临床病理学特征的关系[J]. 诊断学理论与实践, 2017, 16(02): 166-170. |
| [14] | 赵建溪, 任刚, 蔡嵘, 郭辰, 陈健, 李华莉,. 多排螺旋CT诊断早期胃癌淋巴结转移的准确性研究[J]. 诊断学理论与实践, 2016, 15(02): 174-179. |
| [15] | 贺文广, 任刚,. 胃癌淋巴结转移规律及淋巴结清扫范围的判定[J]. 诊断学理论与实践, 2016, 15(01): 65-68. |
| 阅读次数 | ||||||
|
全文 |
|
|||||
|
摘要 |
|
|||||